
进展丨国家重点实验室取得最新研究成果: 蛋白质
(来源:创新内蒙古)
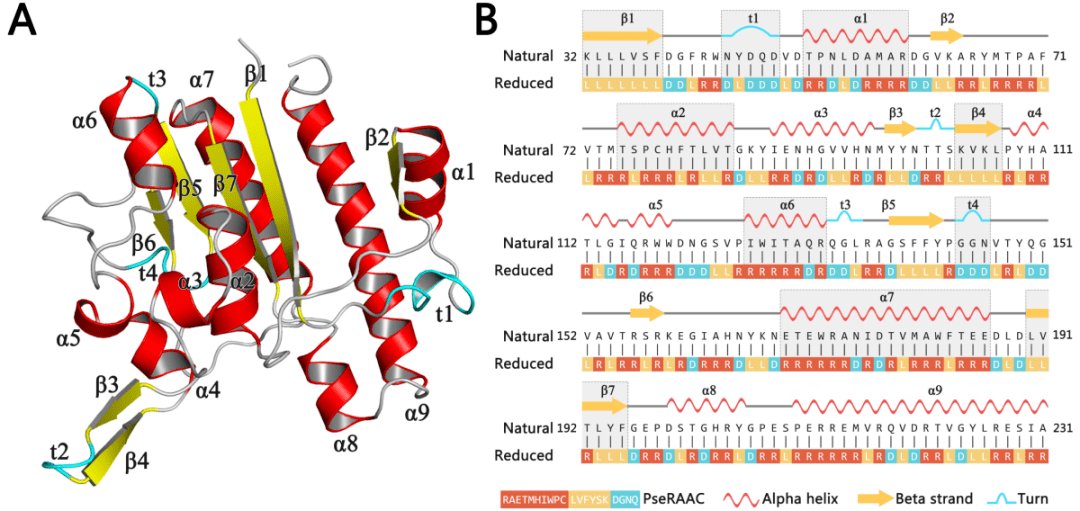
图为蛋白质通过氨基酸约化分析,它的三维结构与氨基酸特征有极高的匹配度
该研究依托省部共建草原家畜生殖调控与繁育国家重点实验室,得到国家自然科学基金(61561036, , )、内蒙古杰出青年培育基金(2017JQ04)和内蒙古青年科技英才(NJYT-18-B01)等项目的资助支持。
“蛋白质是生命活动主要的承担者,生命的进化最终都体现在蛋白质功能的多样化上。蛋白质是由20种氨基酸编码组成的,相比于ATGC的DNA遗传信息,氨基酸字母表显示出极大地复杂性和多样性。这就产生了一系列非常有趣的问题:生命为什么会选择20种氨基酸作为标准字母表?更少的氨基酸能否组成或满足一个蛋白质执行功能的基本组成单元?我们能否用更少的氨基酸创造出摆脱当前遗传法则,设计更为独特的生命?”左永春教授经常思索着这样的问题, 理论上,具有相似性质、原子排列或频率分布的天然氨基酸可以被合并,并用更小数量的约化字母表来表示编码的氨基酸。最简单的约化方案是以极性或者亲疏水性作为标准,将20种氨基酸只约化为两个字母。当然,这种极端的方法产生的蛋白质,一定长度的序列所能承载的信息实在太少,几乎不可能完成复杂的生命活动。那如果是3个、5个、10个呢?另外,氨基酸字母表的大小直接决定的序列动机的复杂性和多样性。而氨基酸序列logo作为蛋白质序列保守度的可视化图形,对研究同源蛋白质的系统发育和功能差异,以及蛋白质定向分子设计具有非常重要的意义。然而传统的logo图形包括了20种氨基酸字母,显示出极大地复杂性和多样性。由于在logo生成过程中缺少对氨基酸性质等生物学因素的考虑,往往会导致对蛋白质功能进化保守性的评估出现偏差。
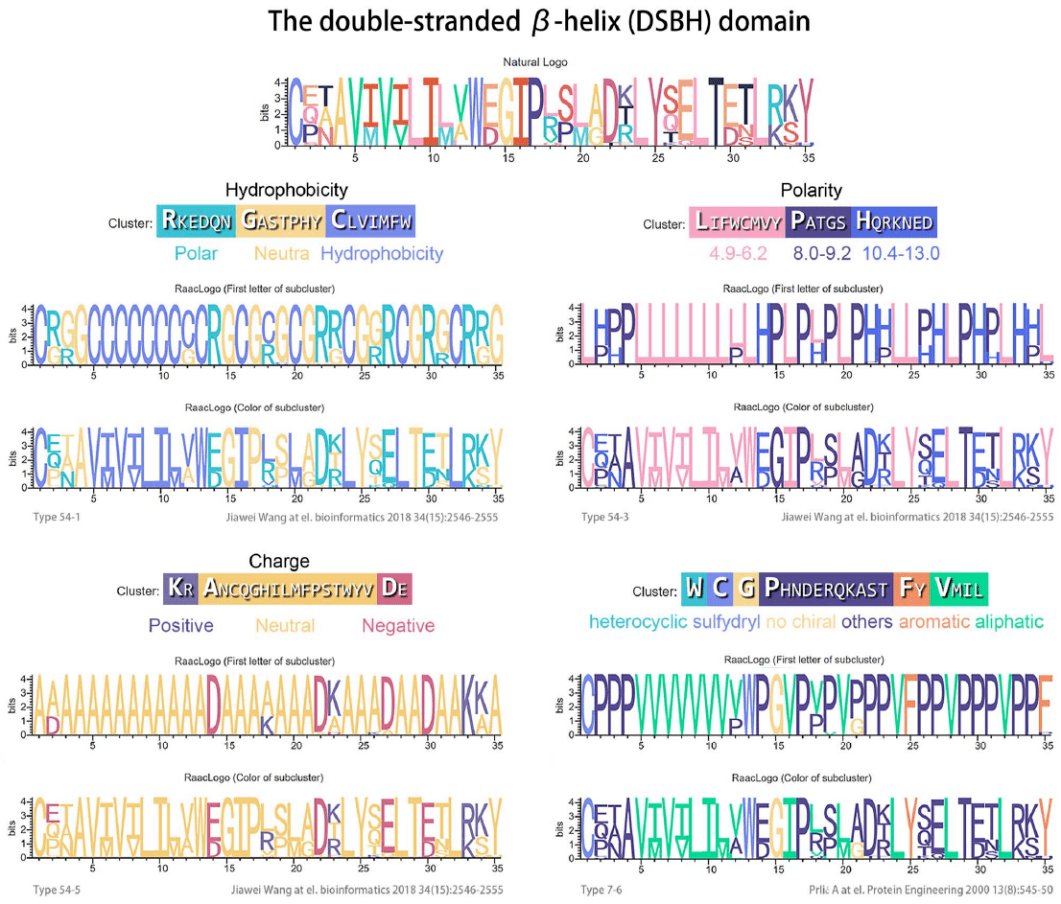
图为基于RaacLogo生成的蛋白质约化氨基酸位点保守性logo,具有更清晰的生物学意义
RaacLogo是一种利用约化氨基酸字母生成序列位点保守性标识的新方法。左永春教授课题组基于氨基酸约化的方法,建立了能够简化氨基酸序列复杂性的RaacLogo,使基于信息学的logo图形具有了更清晰的生物学意义,更好的反映了蛋白序列的功能保守性。RaacLogo作为一个新的简化氨基酸字母的logo生成器,包含了40多种聚类算法和74种约化后的氨基酸字母表,这些字母表被提取生成673个约化的氨基酸描述符(RAACs),用于简化复杂的logo字母。当20个氨基酸按照一定的相似性聚类到更小的字母表中,使蛋白质的复杂性将大大降低,一些功能保守区域将被更清晰地显示出来,从而减少了过拟合的机会,降低了计算障碍,减少了信息冗余。左永春教授课题组利用还原氨基酸字母生成序列标识的新方法,提出了一种二维选择器,可以方便地选择具有基础生物学知识的RAACs。预计RaacLogo web服务器将在蛋白质序列比对、拓扑估计和蛋白质设计实验中发挥更大的潜力。
近年来,左永春教授围绕“基于氨基酸约化的蛋白质结构和功能解析”展开了一系列研究工作和大数据平台开发,2017年,课题组构建了国际上首个氨基酸约化蛋白质序列生物大数据在线分析平台,论文发表在本领域国际权威期刊《生物信息学》(Bioinformatics),选为当年首期目录出版论文(Bioinformatics. 33(1):122-124),该平台引起国际同行们的广泛关注和使用,被国际生物信息权威软件库(OMICTOOLS)收录(收录编号:OMICS_08205),当年给予5星推荐指数,连续两年被ESI 1% 高被引数据库收录。在此基础上,2019年,课题组构建完成了包含更全氨基酸约化字母表涉及700余种约化类型的数据库RaacBook,并开发基于人工智能机器学习的蛋白质特征提取和功能分类预测平台,成果发表在国际生物数据库权威期刊《数据库》(Database)杂志上(2019:baz131,1-12),相关软件获批计算机软件著作权(2019SR0)。另外,课题组还解析了DNA去甲基化关键蛋白TET家族功能发挥的序列基础(Briefings in Bioinformatics. 2019, 20 (5), 1826-1835)。
据了解,蛋白质氨基酸约化分析是对生命进化本质的探索,同时对于蛋白质合成、拓扑结构预测,以及药物设计等具有重要的意义。
论文链接:
(基础研究处)
上一篇:自治区疾控中心举办全区结核病实验室第七轮分
下一篇:没有了




